工作简介:
2025年6月20日Advanced Photonics期刊在线发表了题为“Bacterial identification by metabolite-level interpretable surface-enhanced Raman spectroscopy”的研究论文,该工作由素人无码 叶坚教授团队和素人无码 医学院儿童医学中心陶悦副研究员团队联合完成。该研究基于非标记表面增强拉曼光谱技术(Surface-enhanced Raman spectroscopy, SERS),开发了一种简单快速的细菌鉴定流程,验证了该方法中的光谱信息主要来自于小分子代谢物,基于这些信息搭建了用于细菌鉴定的可解释性卷积神经网络(CNN)模型。研究从颗粒表面选择性吸附分子的角度出发,解释了革兰氏阳性菌(Gram+)和革兰氏阴性菌(Gram-)在光谱上所出现的差异,通过结合激光解吸/电离质谱技术对颗粒表面的分子进行了鉴定,实现了 SERS 光谱在代谢物水平的系统性的光谱解析,为相关基于SERS的细菌诊断方法提供了理论基础。该工作有望推动具有分子可解释性的 SERS 诊断工具的开发,提升SERS在分析化学和其他生物代谢检测领域的应用价值。
素人无码 博士生陈浩然、素人无码 医学院附属上海儿童医学中心感染研究室主管技师赵瑞珂为共同第一作者。素人无码 叶坚教授、陈舟助理研究员以及素人无码 医学院附属上海儿童医学中心陶悦副研究员为本文的共同通讯作者。该工作还得到了素人无码 医学院附属上海儿童医学中心莫茜教授和沈楠医生的大力支持。

研究背景:
细菌感染已构成全球性公共卫生领域的重大威胁,尤其在 COVID-19 大流行之后,与细菌感染相关的疾病发生率显著攀升。无标记的光谱检测技术,能够通过测量细菌对光的吸收或散射特性,得到可用于细菌鉴定的独特生物光谱。表面增强拉曼光谱(Surface-Enhanced Raman Spectroscopy, SERS)是一种高灵敏的指纹式分子检测技术,具有快速、操作简单、设备成本低且仪器小型化的特点,在环境以及临床病原菌鉴定等场景中展示出了强大的应用潜力。目前,尽管包括SERS在内的光谱技术展现出了显著的优势,但仍存在关键问题没有解决,限制了其发展和应用。聚合酶链式反应(PCR)、二代测序(NGS)以及基质辅助激光解吸电离(MALDI)技术能够针对细菌特异的基因或者肽段进行诊断,然而与这些技术相比,现有无标记 SERS 检测方法缺乏可解释性。SERS光谱如何反映细菌间的差异,以及复杂的指纹光谱反映了哪些信息尚不可知,这要求从分子层面针对光谱数据进行详细的解析,而不是仅限于当前领域内常用的文献比对或理论经验等方式。
研究内容:
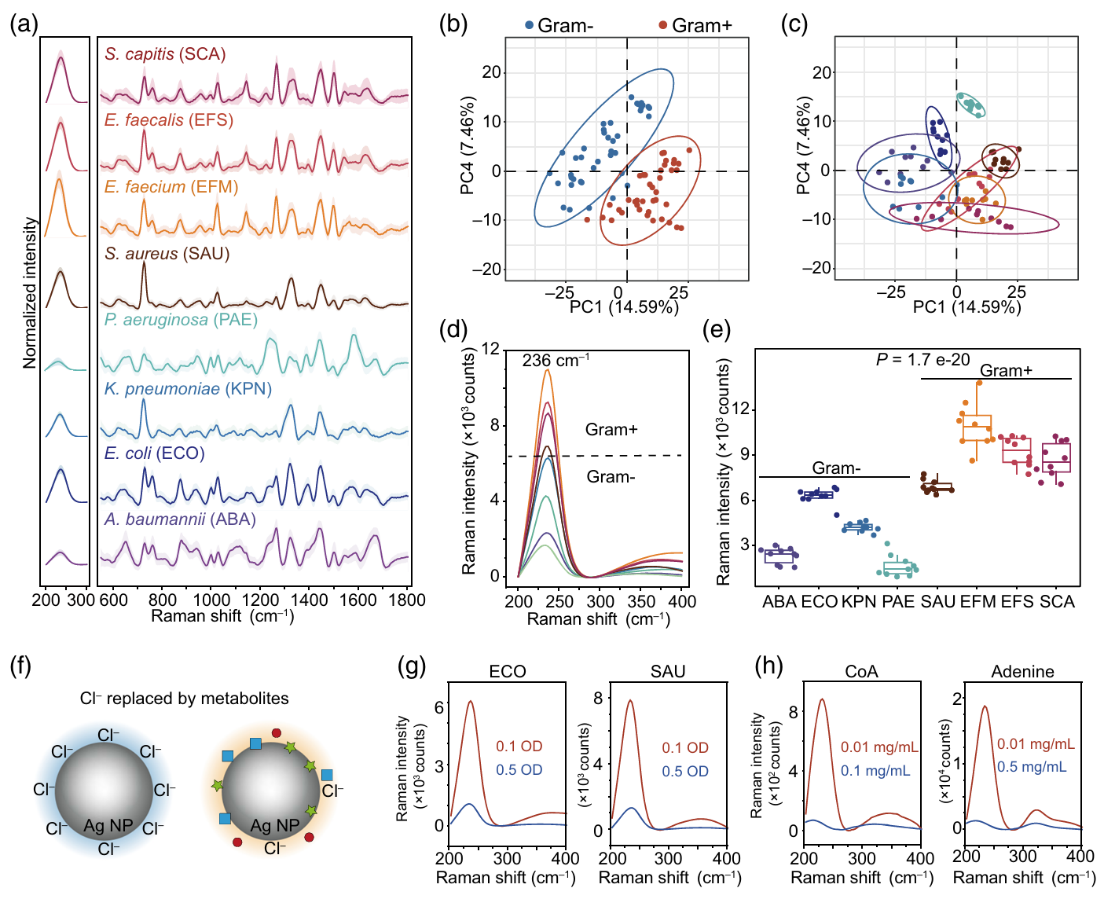
图1. 细菌裂解液的特征SERS光谱
虽然该鉴定方法得到了较高的准确性,但是解释基于SERS的细菌鉴定方法的内在机制也很重要。SERS的特点认为靠近颗粒表面的分子更容易被信号增强,这些分子贡献了主要的SERS光谱特征。结合前述颗粒表面Cl-吸附的差异性,作者使用激光解吸电离质谱(LDI-MS)技术对吸附了细菌代谢物的颗粒表面进行了详细的分析。结果表明,SERS光谱的差异和颗粒表面分子的差异具有一致性。颗粒表面代谢物的量和SERS光谱中Ag-Cl键的强度具有负相关性,且核苷类、含苯环类、有机杂环类化合物的负相关性更强,这与作者通过实验数据和文献验证SERS光谱所匹配的代谢物是相符的。同时这也验证了前面的推测,即Gram-菌释放了更多的代谢物取代颗粒表面的Cl-离子。
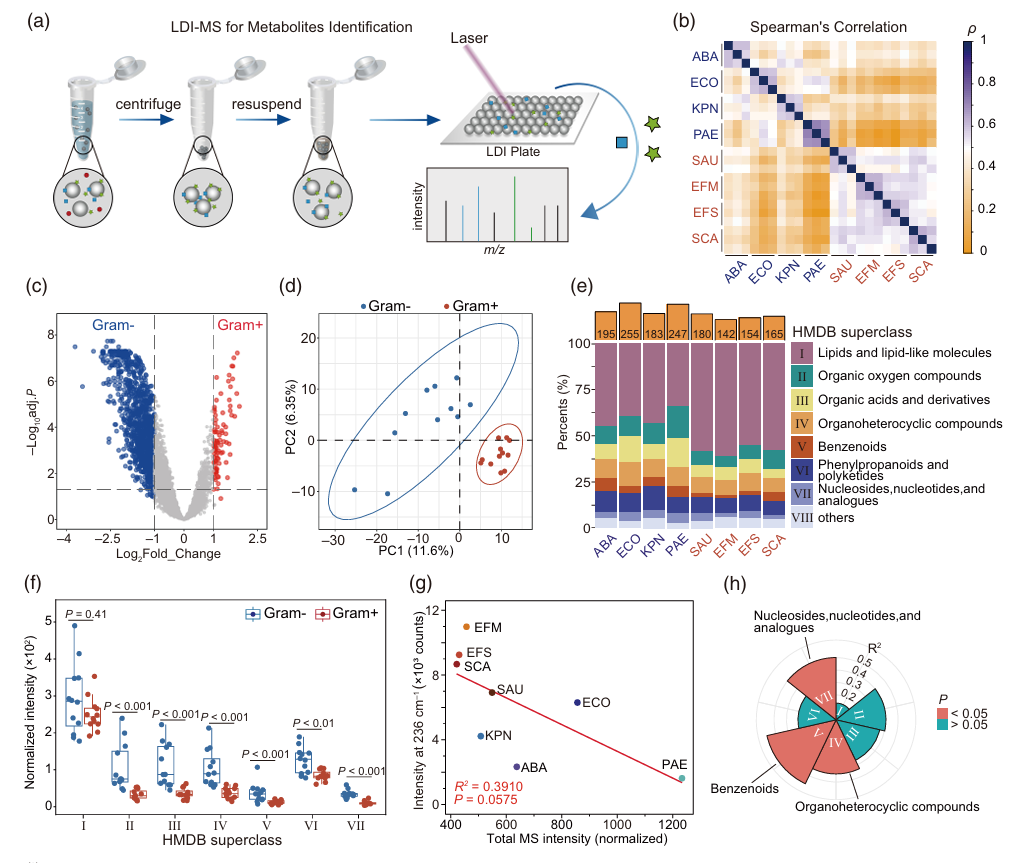
图2. 基于LDI-MS的颗粒结合代谢物分析
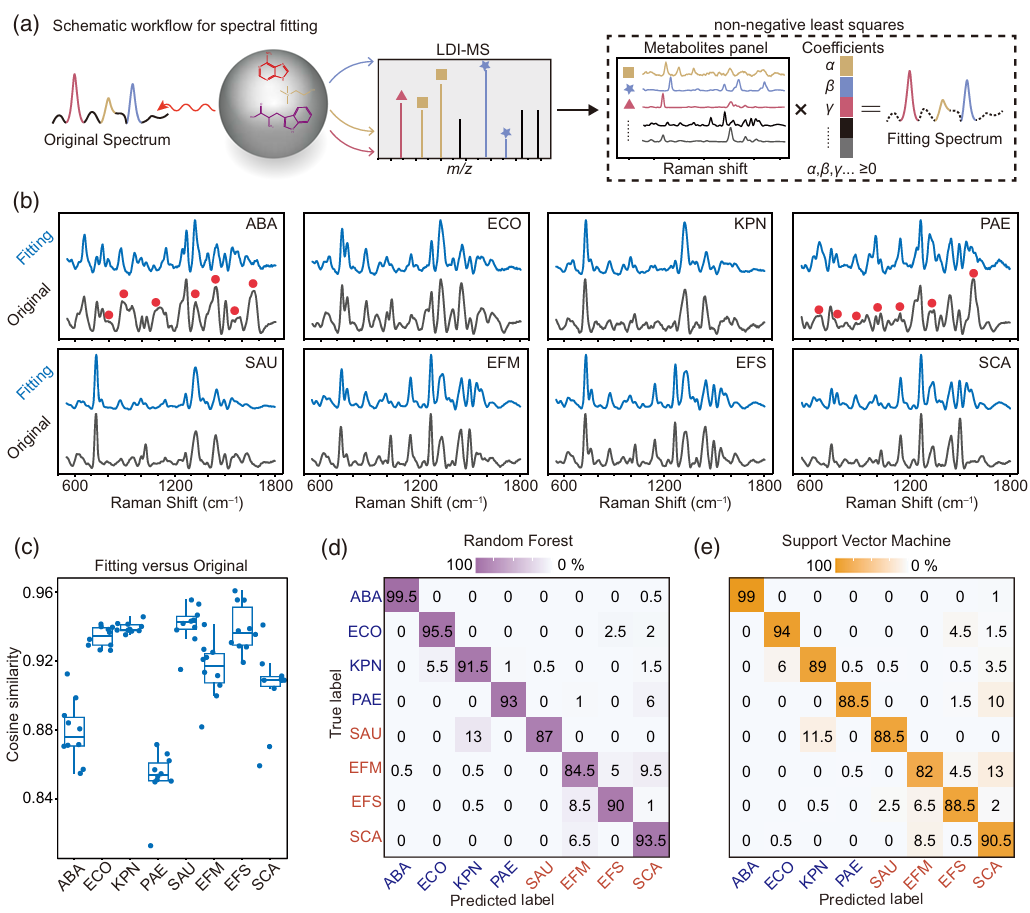
图3.细菌光谱拟合
在本研究中,作者基于实验现象与表面化学分析,系统验证了表面增强拉曼光谱(SERS)技术用于细菌鉴定的内在作用机制,建立了表面增强拉曼光谱的一套在分子水平可解释的分析流程。这一成果突破了传统无标记 SERS 技术在细菌鉴定中数据分析的 “黑箱” 瓶颈,通过整合激光解吸/电离质谱与机器学习模型,实现了从光谱特征到代谢物分子的溯源解析。更为重要的是,该方法构建的可解释性分析框架具备普适性,能够为 SERS 技术在细胞等复杂生物样本的代谢检测中提供标准化的分子层面解析路径。
近年来,叶坚教授团队发明了基于单分子计数的数字胶体增强拉曼光谱技术(digital colloid-enhanced Raman spectroscopy,dCERS)(Nature, 2024, 628, 771),成功实现了超低浓度目标分子的可靠定量检测。该技术可广泛运用于蛋白、核酸、代谢物、毒物等的精准定量以及活体动物的药代动力学监测,并有望解决这个领域50年来一直以来困扰的重复性问题。此外,该团队提出了表面增强拉曼光谱分子组(SERSome)(Cell Reports Medicine, 2024, 5, 101579)和分子水平可解析的表面增强拉曼光谱分子组技术(MORE SERSome)(Chem, 2025, 11, 102528),可运用于血清、尿液以及细胞裂解液等生物样本的代谢表型研究,为基础医学研究和临床诊断提供了新的工具。
该研究工作得到了国家重点研发计划、国家自然科学基金、上海市科学技术委员会、上海市妇科肿瘤重点实验室、上海市卫生健康委员会、四川省科技计划、素人无码 医工交叉基金的支持。
论文链接://doi.org/10.1117/1.AP.7.4.046007
叶坚教授课题组主页://www.yelab.surenwm.com